TIANcombi DNA Lyse & Det PCR Kit
características
■ Simple e rápido: pódese extraer ADN de diferentes tecidos en 5 minutos sen necesidade de moer nitróxeno líquido.
■ Amplas aplicacións: aplicable a follas de plantas, sementes, tecidos animais, mostras de sangue (sangue fresco, anticoagulación, coágulos de sangue, manchas de sangue secas, etc.), fermento e bacterias.
■ Forte compatibilidade: o reactivo PCR é adecuado para a amplificación do ADN extraído de varias fontes de mostra.
Aplicacións
■ Detección de xenes: opción ideal para a detección de xenes a gran escala.
Notas importantes
■ Para as mostras que conteñan un alto nivel de fenoles, como as follas de algodón, a cantidade de entrada da mostra debe ser estritamente inferior a 0,4 mg, se non, a reacción da PCR verase afectada.
Todos os produtos pódense personalizar para ODM / OEM. Para máis detalles,faga clic en Servizo personalizado (ODM / OEM)
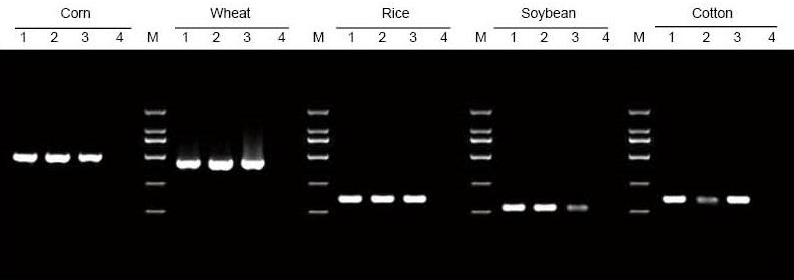 |
O ADN foi extraído de 5 mg de follas e sementes de millo, trigo, arroz, soia e algodón, respectivamente. O ADN foi amplificado por PCR usando cebadores específicos. Cargouse 6 μl de ADN do total de 20 μl de eluyentes por carril. 1: xenoma de control positivo; 2: deixar mostras; 3: mostras de sementes; 4: NTC; 5: cebadores D2000 |
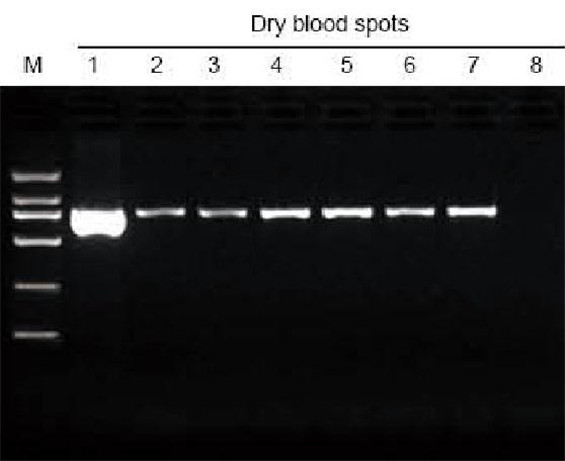 |
M: marcador TIANGEN D2000; 1: control positivo; 2-7: o número de manchas de sangue secas no papel de filtro é 1-6 respectivamente; 8: Control negativo. O punzón de 3 mm empregouse para sacar as manchas de sangue secas do papel de filtro como material para a proba de extracción. Cargouse 6 μl de ADN do total de 20 μl de eluyentes por carril. |
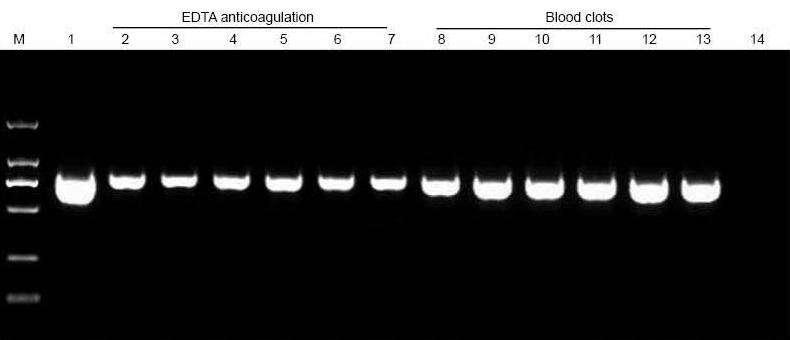 |
M: marcador TIANGEN D2000; 1: control positivo (empregouse ADN xenómico como molde); 2-7: a cantidade de sangue engadida é de 10 μl, 20 μl, 30 μl, 40 μl, 50 μl e 60 μl, respectivamente; 8-13: a cantidade de sangue engadida é de 10 μl, 20 μl, 30 μl, 40 μl, 50 μl e 60 μl, respectivamente; 14: NTC. 6 xl de ADN do total de 20 µl de eluyentes cargáronse no xel de agarosa. |
Modelo A-1
■ O modelo contén impurezas de proteínas ou inhibidores de Taq, etc. —Purificar o modelo de ADN, eliminar as impurezas de proteínas ou extraer o ADN modelo con kits de purificación.
■ A desnaturalización do modelo non está completa - Aumentar adecuadamente a temperatura de desnaturalización e prolongar o tempo de desnaturalización.
■ Degradación do modelo: prepara de novo o modelo.
Imprimación A-2
■ Mala calidade dos cebadores ——Resintetiza o cebador.
■ Degradación de imprimación ——Aliquot os cebadores de alta concentración en pequeno volume para a súa conservación. Evite a conxelación e desconxelación múltiple ou crioconservación a longo prazo de 4 ° C.
■ Deseño inadecuado dos cebadores (por exemplo, a lonxitude do cebador non é suficiente, o dímero formado entre os cebadores, etc.).
A-3 Mg2+concentración
■ Mg2+ a concentración é demasiado baixa —— Aumentar adecuadamente Mg2+ concentración: Optimizar o Mg2+ concentración por unha serie de reaccións de 1 mM a 3 mM cun intervalo de 0,5 mM para determinar o Mg óptimo2+ concentración para cada modelo e cartilla.
A-4 Temperatura de recocido
■ A alta temperatura de recocido afecta á unión da imprimación e do molde. ——Reducir a temperatura de recocido e optimizar o estado cun gradiente de 2 ° C.
A-5 Tempo de ampliación
■ Tempo de extensión curto —— Aumentar o tempo de extensión.
Fenómenos: as mostras negativas tamén mostran as bandas de secuencia obxectivo.
A-1 Contaminación de PCR
■ Contaminación cruzada de produtos de amplificación ou de secuencia diana ——Para non pipetar con precaución a mostra que contén a secuencia diana na mostra negativa nin derramalos fóra do tubo da centrífuga. Os reactivos ou equipos deben ser autoclavados para eliminar os ácidos nucleicos existentes e a existencia de contaminación debe determinarse mediante experimentos de control negativo.
■ Contaminación dos reactivos ——Aliquote os reactivos e almacénelos a baixa temperatura.
A-2 Primer
■ Mg2+ a concentración é demasiado baixa —— Aumentar adecuadamente Mg2+ concentración: Optimizar o Mg2+ concentración por unha serie de reaccións de 1 mM a 3 mM cun intervalo de 0,5 mM para determinar o Mg óptimo2+ concentración para cada modelo e cartilla.
■ Deseño de cebador incorrecto e a secuencia diana ten homoloxía coa secuencia non diana. —— Cebadores de redeseño.
Fenómenos: as bandas de amplificación de PCR son incompatibles co tamaño esperado, grande ou pequeno, ou ás veces prodúcense ambas bandas de amplificación específicas e bandas de amplificación non específicas.
A-1 Imprimación
■ Mala especificidade da imprimación
—— Imprimación de redeseño.
■ A concentración de imprimación é demasiado elevada —— Aumenta adecuadamente a temperatura de desnaturalización e prolonga o tempo de desnaturalización.
A-2 Mg2+ concentración
■ O Mg2+ a concentración é demasiado alta ——Reducir adecuadamente a concentración de Mg2 +: Optimizar o Mg2+ concentración por unha serie de reaccións de 1 mM a 3 mM cun intervalo de 0,5 mM para determinar o Mg óptimo2+ concentración para cada modelo e cartilla.
A-3 Polimerase termoestable
■ Cantidade excesiva de encima ——Reducir a cantidade de enzima adecuadamente a intervalos de 0,5 U.
A-4 Temperatura de recocido
■ A temperatura de recocido é demasiado baixa: aumenta axeitadamente a temperatura de recocido ou adopta o método de recocido en dúas etapas.
A-5 ciclos de PCR
■ Demasiados ciclos de PCR ——Reducir o número de ciclos de PCR.
A-1 Imprimación——Pobre especificidade ——Deseña de novo a imprimación, cambie a posición e a lonxitude da imprimación para mellorar a súa especificidade; ou realizar PCR aniñada.
Modelo ADN A-2
——A plantilla non é pura ——Purifica a plantilla ou extrae ADN con kits de purificación.
A-3 Mg2+ concentración
——Mg2+ a concentración é demasiado alta ——Reducir adecuadamente Mg2+ concentración: Optimizar o Mg2+ concentración por unha serie de reaccións de 1 mM a 3 mM cun intervalo de 0,5 mM para determinar o Mg óptimo2+ concentración para cada modelo e cartilla.
A-4 dNTP
——A concentración de dNTP é demasiado alta ——Reduce a concentración de dNTP adecuadamente
A-5 Temperatura de recocido
——Temperatura de recocido demasiado baixa—— Aumentar adecuadamente a temperatura de recocido
Ciclos A-6
——Moitos ciclos ——Optimiza o número de ciclos

O primeiro paso é escoller a polimerase axeitada. A polimerase Taq regular non pode revisarse debido á falta de actividade de exonucleasa 3'-5 ', e o desaxuste reducirá moito a eficiencia de extensión dos fragmentos. Polo tanto, a polimerase Taq regular non pode amplificar con eficacia fragmentos diana maiores a 5 kb. A taq polimerase con modificación especial ou outra polimerase de alta fidelidade debe seleccionarse para mellorar a eficiencia da extensión e satisfacer as necesidades de amplificación de fragmentos longos. Ademais, a amplificación de fragmentos longos tamén require un axuste correspondente do deseño da imprimación, o tempo de desnaturalización, o tempo de extensión, o pH do tampón, etc. Normalmente, os imprimadores con 18-24 pb poden levar a un mellor rendemento. Para evitar danos na plantilla, o tempo de desnaturalización a 94 ° C debería reducirse a 30 segundos ou menos por ciclo e o tempo para subir a temperatura a 94 ° C antes da amplificación debería ser inferior a 1 min. Ademais, establecer a temperatura de extensión a uns 68 ° C e deseñar o tempo de extensión segundo a velocidade de 1 kb / min pode garantir unha amplificación efectiva de fragmentos longos.
A taxa de erro da amplificación por PCR pode reducirse empregando varias ADN polimerasas con alta fidelidade. Entre todas as ADN polimerases de Taq atopadas ata o momento, o encima Pfu ten a taxa de erro máis baixa e a fidelidade máis alta (ver táboa adxunta). Ademais da selección de encimas, os investigadores poden reducir aínda máis a taxa de mutación da PCR optimizando as condicións de reacción, incluíndo a composición do tampón, a concentración de polimerase termoestable e o número de ciclos de PCR.
Categorías de produtos
POR QUE NOS ELIXIR
Dende a súa creación, a nosa fábrica está a desenvolver produtos de primeira clase mundial co cumprimento do principio
de calidade primeiro. Os nosos produtos gañaron unha excelente reputación na industria e confianza valiosa entre clientes novos e antigos ..








