TIANSeq Kit de biblioteca de ADN rápido (iluminación)


características
- Boa uniformidade de secuenciación: sen sesgo de base do proceso de fragmentación do ADN e do proceso de amplificación da PCR.
- Alta eficiencia de conversión de bibliotecas: pódese asegurar a construción de biblioteca de alta eficiencia ata mostras de ADN de 0,25 ng.
- Funcionamento rápido: todo o proceso de preparación da biblioteca só precisa 2 horas.
Especificación
Tipo: Preparación de biblioteca de ADN para a plataforma de secuenciación de alto rendemento de iluminina.
Mostra: ADNc ou pequenos fragmentos de ADN despois do corte mediante tratamento ultrasónico ou encimático.
Obxectivo: ADN bicatenario.
Entrada de mostra inicial: 0,25 ng- 1 μg.
Tempo de operación: 2-2,5 horas.
Aplicacións downstream: Secuenciación na plataforma de iluminación.
s
Todos os produtos pódense personalizar para ODM / OEM. Para máis detalles,faga clic en Servizo personalizado (ODM / OEM)

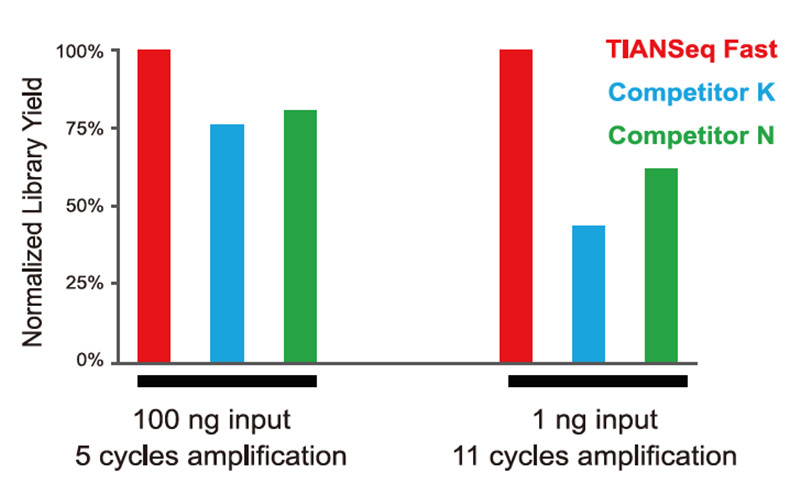 |
Comparación do rendemento da construción da biblioteca Figura 1. Comparación do rendemento da biblioteca entre diferentes produtos |
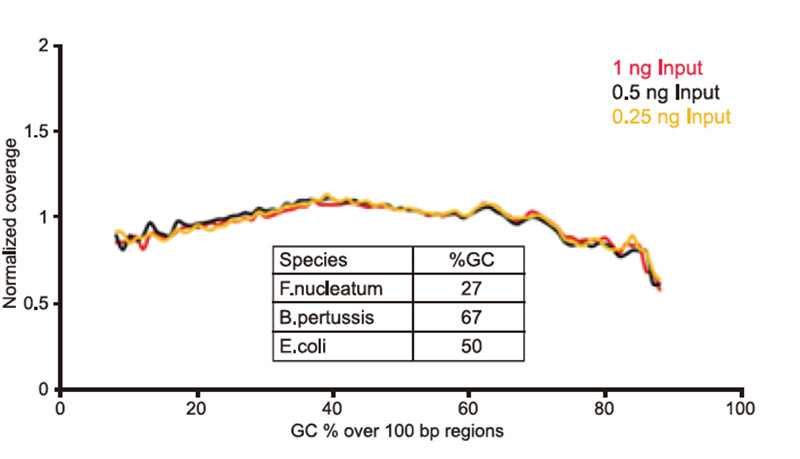 |
Cobertura do xenoma de ADN de baixa entrada Figura 2. Resultados da cobertura do xenoma de ADN de baixa entrada. Comparación das taxas de cobertura de diferentes entradas de ADN (1,0,5,0,25 ng) das tres mesturas de xenomas bacterianos con diferentes contidos de GC na mesma relación molar. Os resultados mostran que o kit de biblioteca de ADN rápido TIANSeq garante unha boa estabilidade e unha alta taxa de conversión para a construción de biblioteca de mostras de traza. |
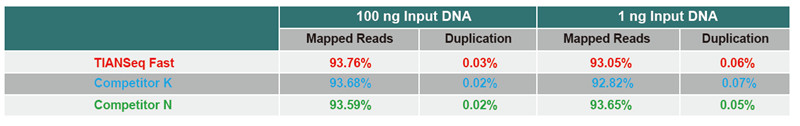 |
Comparación de datos de secuenciación de diferentes produtos |
Actualmente, a tecnoloxía de secuenciación de alto rendemento baséase principalmente na tecnoloxía de secuenciación de próxima xeración. Como a lonxitude de lectura da tecnoloxía de secuenciación de próxima xeración é limitada, debemos dividir a secuencia de lonxitude completa en pequenas bibliotecas de fragmentos para secuencialas. Segundo as necesidades de diferentes experimentos de secuenciación, normalmente escollemos unha secuenciación simple ou dobre. Actualmente os fragmentos de ADN da biblioteca de secuenciación de próxima xeración distribúense polo xeral entre 200 e 800 pb.

a) O ADN é de baixa calidade e contén inhibidores. Use mostras de ADN de alta calidade para evitar a inhibición da actividade enzimática.
b) A cantidade de mostra de ADN é insuficiente cando se usa un método sen PCR para construír unha biblioteca de ADN. Cando a entrada do ADN fragmentado supera os 50 ng, pódese realizar selectivamente un fluxo de traballo sen PCR durante o proceso de construción da biblioteca. Se o número de copias da biblioteca é demasiado baixo para ser secuenciado directamente, a biblioteca de ADN pode amplificarse mediante PCR despois da ligadura do adaptador.
c) A contaminación do ARN leva a unha cuantificación inicial do ADN imprecisa Pode existir contaminación do ARN no proceso de purificación do ADN xenómico, o que pode provocar unha cuantificación do ADN imprecisa e unha carga de ADN insuficiente durante a construción da biblioteca. O ARN pode eliminarse tratándose con RNase.
A-1
a) Aparecen fragmentos pequenos (60 pb-120 pb) Os fragmentos pequenos adoitan ser fragmentos de adaptadores ou dímeros formados por adaptadores. A purificación con contas magnéticas Agencourt AMPure XP pode eliminar de forma eficaz estes fragmentos de adaptador e garantir a calidade da secuenciación.
b) Os fragmentos grandes aparecen na biblioteca despois da amplificación por PCR. O tamaño do fragmento de ADN da biblioteca aumentará en 120 pb despois de que o adaptador estea ligado. Se o fragmento de ADN aumenta máis de 120 pb despois da ligadura do adaptador, pode ser causado por unha amplificación anormal do fragmento dunha amplificación PCR excesiva. A redución do número de ciclos de PCR pode evitar a situación.
c) Tamaño anormal dos fragmentos de ADN da biblioteca despois da ligadura do adaptador A lonxitude do adaptador deste kit é de 60 pb. Cando os dous extremos do fragmento están ligados aos adaptadores, a lonxitude só aumentará 120 pb. Cando use un adaptador distinto ao proporcionado por este kit, póñase en contacto co provedor para proporcionar información relevante como a lonxitude do adaptador. Asegúrese de que o fluxo de traballo e a operación do experimento sigan os pasos descritos no manual.
d) Tamaño anormal do fragmento de ADN antes da ligadura do adaptador A razón deste problema pode ser causada por condicións de reacción incorrectas durante a fragmentación do ADN. Débense usar tempos de reacción diferentes para a entrada de ADN diferente. Se a entrada de ADN é superior a 10 ng, recomendamos escoller o tempo de reacción de 12 min como tempo de inicio para a optimización e o tamaño do fragmento producido neste momento está principalmente no rango de 300-500 pb. Os usuarios poden aumentar ou diminuír a lonxitude dos fragmentos de ADN durante 2-4 minutos segundo os seus propios requisitos para optimizar os fragmentos de ADN co tamaño requirido.
A-2
a) O tempo de fragmentación non está optimizado Se o ADN fragmentado é demasiado pequeno ou demasiado grande, consulte as Directrices para a selección do tempo de fragmentación proporcionadas na instrución para determinar o tempo de reacción e use este punto de tempo como control, configure ademais un sistema de reacción para prolongar ou acurtar 3 min para facer un axuste máis preciso no tempo de fragmentación.
A-3
Distribución anormal do tamaño do ADN despois do tratamento de fragmentación
a) Método incorrecto de desconxelación do reactivo de fragmentación ou o reactivo non está completamente mesturado despois do desconxelamento. Desconxele o reactivo 5 × Fragmentation Enzyme Mix no xeo. Unha vez desconxelado, mestura o reactivo uniformemente batendo suavemente o fondo do tubo. Non revolva o reactivo.
b) A mostra de entrada de ADN contén EDTA ou outros contaminantes O esgotamento de ións de sal e axentes quelantes na etapa de purificación do ADN é particularmente importante para o éxito do experimento. Se o ADN disólvese en 1 × TE, use o método fornecido na instrución para realizar a fragmentación. Se a concentración de EDTA na solución é incerta, recoméndase purificar o ADN e disolvelo en auga desionizada para a súa posterior reacción.
c) Cuantificación inicial do ADN imprecisa O tamaño do ADN fragmentado está intimamente relacionado coa cantidade de entrada de ADN. Antes do tratamento de fragmentación, a cuantificación precisa do ADN usando Qubit, Picogreen e outros métodos é esencial para determinar a cantidade exacta de ADN no sistema de reacción.
d) A preparación do sistema de reacción non segue as instrucións. A preparación do sistema de reacción fragmentado debe realizarse sobre xeo estritamente segundo as instrucións. Para garantir o mellor efecto, todos os compoñentes da reacción deben colocarse sobre xeo e a preparación do sistema de reacción debe realizarse despois do arrefriamento completo. Despois de completar a preparación, toque ou pipetee para mesturar ben. Non vórtice!
1. Un método de mestura inadecuado (vórtice, oscilación violenta, etc.) causará unha distribución anormal dos fragmentos da biblioteca (como se mostra na seguinte figura), afectando así á calidade da biblioteca. Polo tanto, cando prepare a solución de reacción de Fragmentation Mix, pipetee suavemente cara arriba e abaixo para mesturar ou use a punta dos dedos para tocar e mesturar uniformemente. Ten coidado de non mesturarte con vórtice.

2. Para a construción de bibliotecas débese empregar ADN de alta pureza
■ Boa integridade do ADN: a banda de electroforese ten máis de 30 kb, sen colas
■ OD260 / 230:> 1,5
■ OD260 / 280: 1.7-1.9
3. A cantidade de entrada de ADN debe ser precisa. Suxírese empregar métodos Qubit e PicoGreen para cuantificar o ADN, en lugar de Nanodrop.
4. Débese determinar o contido de EDTA na solución de ADN. A EDTA ten unha grande influencia na reacción de fragmentación. Se o contido de EDTA é alto, a purificación do ADN debe realizarse antes da proba posterior.
5. A solución de reacción de fragmentación debe prepararse sobre xeo. O proceso de fragmentación é sensible á temperatura e ao tempo de reacción (especialmente despois de engadir potenciador). Para garantir a precisión do tempo de reacción, prepare o sistema de reacción sobre xeo.
6. O tempo de reacción de fragmentación debe ser preciso. O tempo de reacción do paso de fragmentación afectará directamente ao tamaño dos produtos do fragmento, afectando así a distribución do tamaño dos fragmentos de ADN na biblioteca.
1. Que tipo de mostra é aplicable a este kit?
O tipo de mostra aplicable deste kit pode ser ARN total ou ARNm purificado cunha boa integridade de ARN. Se se usa ARN total para construír a biblioteca, recoméndase usar o kit de esgotamento de ARNr (Cat # 4992363/4992364/4992391) para eliminar primeiro o ARNr.
2. ¿Pódense usar mostras FFPE para construír biblioteca con este kit?
O ARNm nas mostras de FFPE degradarase ata certo punto, cunha relativa integridade deficiente. Ao usar este kit para a construción da biblioteca, recoméndase optimizar o tempo de fragmentación (acurtar o tempo de fragmentación ou non realizar fragmentación).
3. Usando o paso de selección de tamaño indicado no manual do produto, que pode provocar que o segmento inserido presente unha pequena desviación?
A selección do tamaño realizarase de acordo co estricto paso da selección do tamaño deste manual do produto. Se hai desviación, a razón pode ser que as contas magnéticas non están equilibradas á temperatura ambiente ou non están completamente mesturadas, a pipeta non é precisa ou o líquido queda na punta. Recoméndase usar os consellos con baixa adsorción para o experimento.
4. Selección de adaptadores na construción de bibliotecas
O kit de construción da biblioteca non contén reactivo adaptador e recoméndase usar este kit xunto co adaptador de índice único TIANSeq (Illumina) (4992641/4992642/4992378).
5. QC da biblioteca
Detección cuantitativa da biblioteca: Qubit e qPCR úsanse para determinar a concentración de masa e a concentración molar da biblioteca respectivamente. A operación está estrictamente de acordo co manual do produto. A concentración da biblioteca xeralmente cumprirá os requisitos da secuenciación NGS. Detección do rango de distribución da biblioteca: usando Agilent 2100 Bioanalyzer para detectar o rango de distribución da biblioteca.
6. Selección do número do ciclo de amplificación
Segundo as instrucións, o número de ciclos de PCR é de 6-12 e o número de ciclos de PCR necesarios debería seleccionarse segundo a entrada de mostra. Nas bibliotecas de alto rendemento, a sobreamplificación adoita producirse en graos variables, o que se manifesta por un pico lixeiramente maior despois do pico do rango obxectivo na detección do Bioanalizador Agilent 2100, ou a concentración detectada de Qubit é inferior á de qPCR. A amplificación suave é un fenómeno normal, que non afecta a secuenciación da biblioteca e a análise de datos posterior.
7. Spikes aparece no perfil de detección de Agilent 2100 Bioanalyzer
A aparición de picos na detección de Bioanalizador Agilent 2100 débese á fragmentación desigual das mostras, onde haberá máis fragmentos de certo tamaño, e isto farase máis evidente despois do enriquecemento con PCR. Neste caso, suxírese non realizar a selección do tamaño, é dicir, establecer a condición de fragmentación a 94 ° C durante 15 min incubados, onde a distribución do fragmento é pequena e concentrada e a homoxeneidade pode mellorarse.
Categorías de produtos
POR QUE NOS ELIXIR
Dende a súa creación, a nosa fábrica está a desenvolver produtos de primeira clase mundial co cumprimento do principio
de calidade primeiro. Os nosos produtos gañaron unha excelente reputación na industria e confianza valiosa entre os clientes novos e antigos ..










